
Nicolas Durand
-
Compteur de contenus
16 -
Inscription
-
Dernière visite
-
Last Connexion
Soon available - 84317
Messages posté(e)s par Nicolas Durand
-
-
Il y a 9 heures, esoub83 a dit :Je ne sais pas si dans la logique Demetra, le spectre final 1D correspond à la somme des spectres 1D issus de chaque pose unitaire ou si le soft additionne les spectres 2D de chaque pose unitaire puis élabore le spectre 1D. Et dans ce cas, où se trouve le fichier fit correspondant aux spectres 2D additionnés ?
Bonjour,
Dans Demetra, c'est les spectres 1D qui sont empilés. On voit dans la liste des étapes que l'empilage vient après l'extraction. (C'est l'extraction qui "extrait" les spectres 1D à partir des spectres 2D)
Si tu vas dans l'étape d'empilage du traitement, tu peux les voir en double-cliquant sur le nom des fichiers.Pour chaque étape du traitement, tu vois à gauche le(s) spectre(s) source(s) utilisé pour le traitement (ici l'étape d'empilage) et à droite le résultat.
Tu peux double-cliquer sur les nom de fichier pour les afficher (-> on voit bien que c'est les spectres 1D qui sont utilisé)ou faire un clic-droit pour l'afficher dans l'afficheur FIT et avoir le chemin complet du fichier. (Note que les fichiers sont tous dans le répertoire de session. Les fichiers intermédiaires calculés par le traitement sont dans un sous répertoire du nom de l'observation)
Nicolas
-
Je réalise que ma précédente réponse est hors sujet !
1- Attention l'image de calibration reste assez bruité pour que l'algorithme fonctionne bien.
2- La liste des raies de référence s'édite sur le bouton en haut à droite de l'outil de calibration :
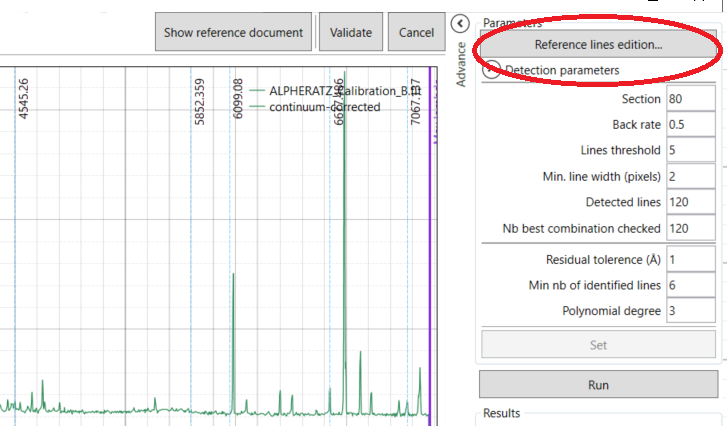
Cordialement,
N. Durand
-
Bonjour
Le choix de la calibration manuelle se fait au niveau de l'étape de calibration : il y a un choix entre "Automatic" et "Manual".
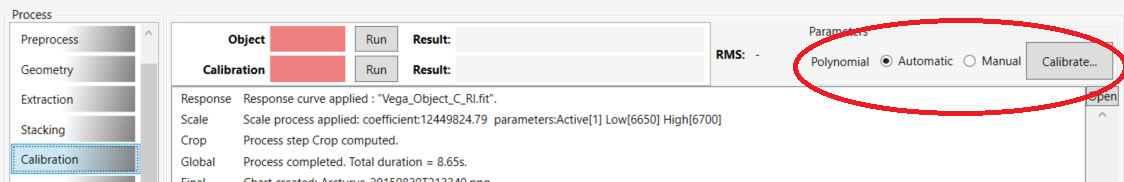
Cordialement,
N. Durand
-
Bonjour,
Le message d'erreur et la correction ne correspondent pas :
- Votre premier message indique un problème de positionnement de l'aire de spectre, malgré votre copie d'écran qui montre que l'aire est bien positionnée.
- Votre 2e message indique qu'après avoir changé les darks (temps de pose différent) le problème a disparu.La seule problématique lié au temps de pose est si la durée d'exposition du Dark est inférieur à la durée d'exposition la plus longue des images des autres paniers.
-> Je suppose que les différentes actions faites ont dû corriger ce qui était à l'origine du 1er problème.
Cordialement
-
Hi,
In addition:
1)I tried the calibration image (vega 2_Calibration_120s_20200729_162339-1.fit) with the calibration tool and the result is OK.

This differs from the "calibration.pdf" screenshot you sent.
I only replaced the calibration image from the Vega observation from the setup with your image (after removing the first line and column to make the image size identical to the other image of the Vega observation)
I didn't changed any process parameter (-> geometry correction is not perfect)
-> The screenshot that may provide other useful informations is this same screen but after clicking on "Detection" radio button near Identification : it allow to see which lines has been detected and first theoretical line that are look.
(or better : share me a wetransfer link with the observation export archive)
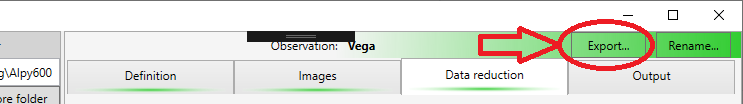
2)
Le 09/08/2020 à 20:48, brownrb1 a dit :Hi Again,
Here's a profile of a spectrum I got last night and when I did the data reduction the same thing happened with 'Geometry' but the wavelength seems to be fairly close.
Dick
I don't think you can check the calibration quality using the target star (Vega) while you change the spectroscope configuration between calibration images acquisition and target star acquisition:
Le 09/08/2020 à 20:24, brownrb1 a dit :I'm not comfortable using the guiding module yet , so I used the small slit to get my bias frames, darks, flats, and calibration and put them in a file. I use the 3mm hole to get my spectra.
-> Applying the detected calibration polynomial function onto the target (star) spectrum image in these condition is not valid. You have to don't change anything on the physical device for it can work. What you are doing here is something "coarse" that can help you for a first try and to help you to understand, learn and go on your learning progress path. But you really need to make the guiding module works to be able to check calibration using the star spectrum.
Nicolas
-
I forgot to post the link about calibration that may help you :
-
Hello Dick
According to your screen, the automatic calibration fail to find all the calibration lines it is looking for.
-> The spectrum may not be "clear" enough (too short exposure, bad focus, ......)
-> The spectrum may not contains the calibration lines that the process is looking for (do you uses the Alpy calibration module ?)
To understand what is going wrong, go to the "Calibration" step and click on "Calibrate..." then "run" then check the "Detection" radio button (under the "run" button on the right side)
-> You will see all detected lines in your spectrum (vertical lines on the spectrum), and you will see the lines list that the process is looking for. Each of theses last lines must be found by the process.
-> Can you post a picture of this screen ?
Nicolas
-
Hello,
sorry for the delay,
- It really look like you created an "eShel" observation and not an alpy one. Can you check the observation process type ? (go to "Definition" tab -> "Context" tab then "Process XXXX" information ? If so, you have to go to session parameters and select "Alpy 600" process then create a new observation.
- Processing calibration step require previous step (stacking) to be already successfully ran. (But if the observation is "eShel", it require the "blaze correction" results, for the selected order, which is impossible in your case (Alpy module)) Is the "stacking" process step successful ?
- About exposures, Demetra considers that every image from the same image basket has the same exposure. It can lead to errors if you merge different exposures images inside the same basket. (I'm not sure to understand "I kept acquired calibration at differents exposures").
If you want to try different exposures, simply copy the observation you made for one exposure and change the image basket for which you want to try another exposure.
- If you are still stuck, can you share me your observation export (right click on the observation -> "export") using something like wetransfert ?
Nicolas
-
Hello,
"eShelCalibX" should be an error reported for eShel process, which is still a Demetra beta version. Can you check if the software process is set to Alpy ? ("Manage observation -> Session parameters -> Context Process" must be Alpy 600)
Can you tell more about what you have done before the error showed up ?
Nicolas
-
Bonjour Frédéric,
1) Tu peux "écraser" Demetra sans problème avec les différents setup existants.
2) Ton fichier comporte des caractères non standard pour du FITS (les 'é' de ton prénom, codé par '233' (probablement lié à un encodage de type "Latin1" (qui est une table ASCII modifiée)) : tu peux le vérifier avec l'outil de la NASA pour vérifier la conformité des fichiers FITS : https://fits.gsfc.nasa.gov/fits_verify.html
Citation1 Header-Data Units in this file.
=================== HDU 1: Primary Array ===================
*** Error: Keyword #9, OBSERVER: String "Frédéric PASTOR" contains non-text characters.
*** Warning: Some CRPIXi keywords appear to be missing; expected 2.
*** Warning: Some CTYPEi keywords appear to be missing; expected 2.
- Demetra ne le lit pas pour une question de compatibilité avec des "vieux" fichiers FITS non conforme, qu'une vieille version de Demetra générait lorsque de tels caractères étaient présents. Cela engendrait un décalage d'octet à rattraper. Rattrapage qui ici, engendre une erreur...
- Je vais modifier Demetra pour qu'il lise quand même ton fichier. Les caractères non conforme seront remplacées par des '?'.
- La prochaine release devrait être disponible dans une semaine ou 2, suivant comment se passe la phase de validation dans laquelle nous sommes actuellement.- En attendant, pour faire fonctionner Demetra, tu n'as pas d'autres choix que d'utiliser des images FITS strictement conformes (sans ces caractères accentués.)
Pour info, le format FITS spécifie une restriction des caractères selon la table ASCII :
Citation4.2.1. Character string
4.2.1.1 Single-record string keywords
A character-string value shall be composed only of the set of
restricted ASCII-text characters, decimal 32 through 126 (hexadecimal
20 through 7E) enclosed by single-quote characters
(“’”, decimal 39, hexadecimal 27). .Nicolas
-
Bonjour Frédéric,
La lecture échoue sur le fichier que tu indiques.
Cela commence par une tentative de décodage d'un mot clé FITS vide... il y a déjà probablement un décalage d'octet à ce moment là, le reste n'est qu'une suite de message découlant de cette première erreur.
Il n'y a rien à configurer dans Demetra pour faire fonctionner les fichiers FITS venant de Prism ou d'ailleurs, et je teste parfois le process Demetra à partir d'images d'utilisateurs acquises avec Prism. Il n'y a pas ce genre d'erreur.
- C'est juste certains fichiers ? as-tu essayé de supprimer seulement le fichier offset-28 du panier des offsets ?
- Quelle version de Demetra utilises-tu ?
- Peux-tu partager un lien https://fromsmash.com/ (ou wetransfer, ou framadrop....) avec ton fichier "C:\Users\LENOVO\Desktop\2020-05-21 Reduction Demetra\offset-28.fits" ?
Merçi
Nicolas
-
Bonjour Vincent,
Oui, c'est un bug côté Demetra que j'ai corrigé hier et qui apparait quand certains mots-clés ne sont pas présents dans l'entête Fits des images acquises.
En attendant un nouveau setup, la seule façon d'activer l'onglet "exploitation" est d'enlever manuellement le 'T' au milieu de la date/heure du nom du fichier résultat du process.
ex : "Vega_20200320T225830.fit" -> "Vega_20200320225830.fit"
Nicolas
-
Bonjour Pierre,
Merçi pour la confirmation.
Attention cependant, la suppression complète de ce répertoire supprime aussi la base de référence spectrale qui n'est installé que par le setup. Si tu as effacé le répertoire complet, il te faut relancer le setup (pas besoin de désinstaller avant) pour la remettre.
Je prépare une nouvelle version corrigeant l'anomalie sous peu.
Nicolas
-
Bonjour,
Je suis le développeur de Demetra, et à l'exemple de vos message, j'ai beaucoup de retour de problème de lancement avec cette version.
Il semble qu'il y ait un problème de lecture du fichier "C:/ProgramData/Demetra/CalibrationParams.xml" pendant le lancement du programme.
En attendant une correction dans le setup, vous pouvez essayer d'effacer ce fichier et de relancer Demetra. (attention, c:\programdata est un répertoire caché). Il sera recréé et devrait s'ouvrir correctement.
Edit: J'ai pu reproduire et corriger l'erreur. Elle apparait si on a préalablement installé une version 4.2.2.0 ou antérieure. Un nouveau setup 5.1.2.0 est prêt. On termine une passe de validation avant de le mettre en ligne.
Nicolas
-
2
-
-
Bonjour Alain,
Non, il n'est pas nécessaire de désinstaller l'ancienne version.
Ce problème apparaît lors de l'installation ou lors du lancement de Demetra après installation ?
Essaye de renommer le répertoire "c:\ProgramData\Demetra" et relancer Demetra.
C'est un répertoire caché par défaut du système d'exploitation.
Nicolas
-
1
-

BeSS - Demetra et rayons cosmiques
dans Spectroscopie et photométrie
Posté(e)
Bonjour Emmanuel,
Non, pas de traitement pour les cosmiques dans Demetra.
Nicolas